因子分析(Factor Analysis) 因子分析 (Factor Analysis)是一种统计方法,旨在通过识别潜在的变量 (因子)来解释观测变量之间的相关性。它广泛应用于心理学、社会科学、市场研究和其他领域,以简化数据结构、减少维度和发现潜在的关系。因子分析 (Factor Analysis)原理是将多个观测变量 归结为少数几个潜在因子。这一过程通常包括以下步骤:1.数据收集与准备 ,收集相关的数据集,确保数据的质量和适用性;2.相关矩阵计算 ,计算观测变量之间的相关矩阵,以了解变量之间的关系;3.因子提取 ,使用统计方法(如主成分分析 或最大似然估计 )提取因子;因子旋转 ,为了使因子更易于解释,通常会对提取的因子进行旋转。旋转方法包括正交旋转 (如Varimax)和斜交旋转 (如Promax);因子解释 ,根据因子的载荷(即每个观测变量与因子的关系)来解释每个因子的含义;模型评估 ,通过各种统计指标(如KMO检验和Bartlett球形检验)评估模型的适用性和有效性。
因子分析的优势:
数据简化 :通过减少变量数量,因子分析可以帮助研究者更好地理解数据结构,降低复杂性。发现潜在结构 :能够识别出影响多个观测变量的潜在因素,从而揭示数据中的隐藏模式。提高模型性能 :在构建预测模型时,使用因子分析提取的因子可以提高模型的准确性和可解释性。
在无监督学习中,有一个数据集高斯分布 的良好特性)是高斯分布 ,因为
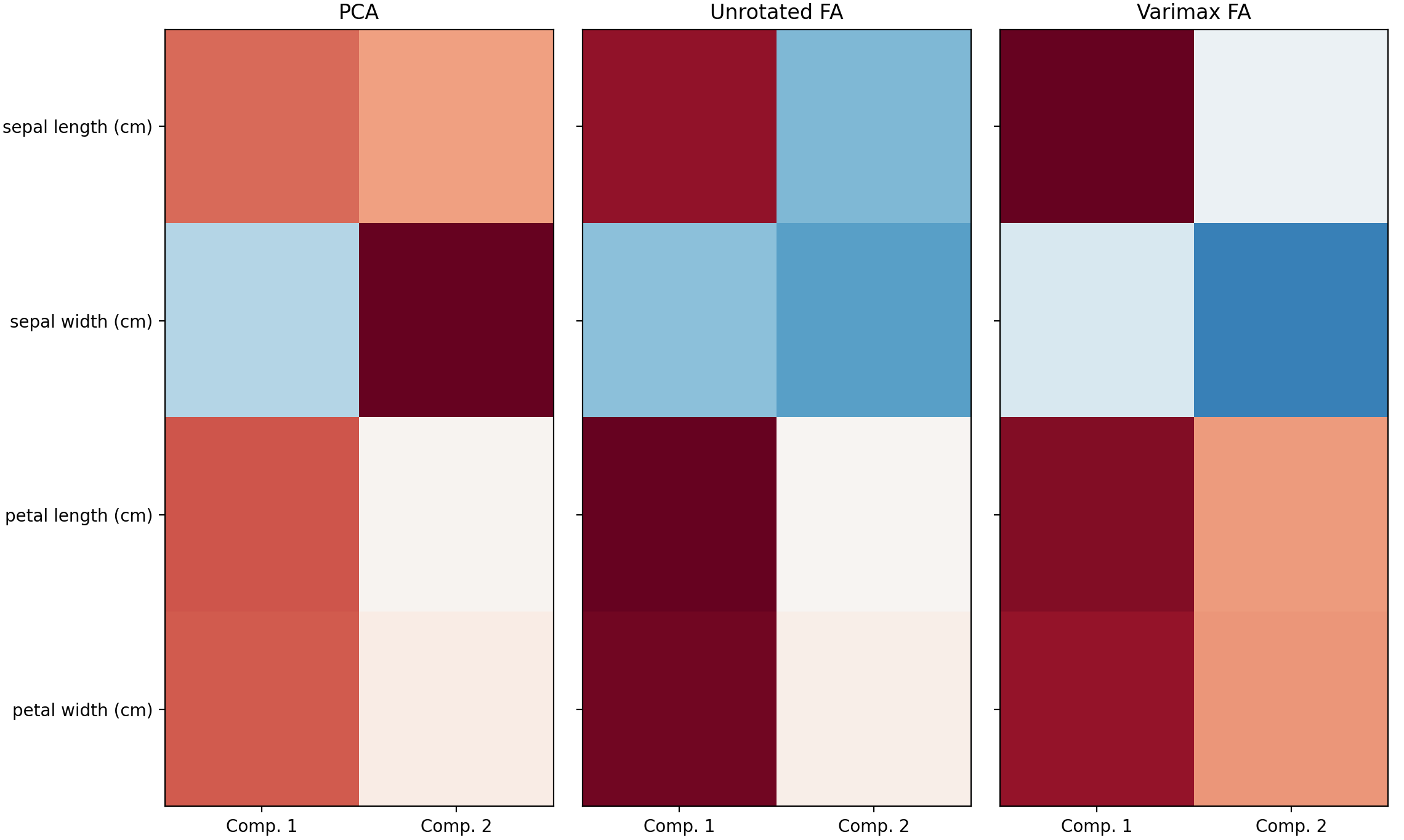
下面的是一个带旋转的因子分析 (Factor Analysis)示例,这里使用鸢尾花数据集,我们发现萼片长度、花瓣长度和宽度与高度相关。矩阵分解技术可以揭示这些潜在模式。对成分进行旋转 并不会提高潜在空间的预测值,但可以在因子分析 (Factor Analysis)中,通过旋转方法(如Varimax旋转),可以优化因子的解释性,以便更清晰地识别出哪些变量与特定因子相关联。在这种情况下,第二个成分的加载值仅在萼片宽度上为正值,从而简化模型并提高可解释性。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 import numpy as npimport matplotlib.pyplot as pltfrom sklearn.datasets import load_irisfrom sklearn.preprocessing import StandardScalerfrom sklearn.decomposition import PCA, FactorAnalysisdata = load_iris() X = StandardScaler().fit_transform(data["data" ]) feature_names = data["feature_names" ] ax = plt.axes() ax.set_xticks([0 , 1 , 2 , 3 ]) ax.set_xticklabels(list (feature_names), rotation=90 ) ax.set_yticks([0 , 1 , 2 , 3 ]) ax.set_yticklabels(list (feature_names)) im = ax.imshow(np.corrcoef(X.T), cmap="RdBu_r" , vmin=-1 , vmax=1 ) plt.colorbar(im).ax.set_ylabel("$r$" , rotation=0 ) ax.set_title("Iris feature correlation matrix" ) plt.tight_layout() n_comps = 2 methods = [ ("PCA" , PCA()), ("Unrotated FA" , FactorAnalysis()), ("Varimax FA" , FactorAnalysis(rotation="varimax" )), ] fig, axes = plt.subplots(ncols=len (methods), figsize=(10 , 8 ), sharey=True ) for ax, (method, fa) in zip (axes, methods): fa.set_params(n_components=n_comps) fa.fit(X) components = fa.components_.T print ("\n\n %s :\n" % method) print (components) vmax = np.abs (components).max () ax.imshow(components, cmap="RdBu_r" , vmax=vmax, vmin=-vmax) ax.set_yticks(np.arange(len (feature_names))) ax.set_yticklabels(feature_names) ax.set_title(str (method)) ax.set_xticks([0 , 1 ]) ax.set_xticklabels(["Comp. 1" , "Comp. 2" ]) fig.suptitle("Factors" ) plt.tight_layout() plt.show()
输出结果为:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 PCA : [[ 0.52106591 0.37741762] [-0.26934744 0.92329566] [ 0.5804131 0.02449161] [ 0.56485654 0.06694199]] Unrotated FA : [[ 0.88096009 -0.4472869 ] [-0.41691605 -0.55390036] [ 0.99918858 0.01915283] [ 0.96228895 0.05840206]] Varimax FA : [[ 0.98633022 -0.05752333] [-0.16052385 -0.67443065] [ 0.90809432 0.41726413] [ 0.85857475 0.43847489]]
概率主成分分析 (PPCA)是一种基于概率模型 的降维技术,旨在通过潜在变量模型来分析数据。与传统的主成分分析 (PCA)不同,概率主成分分析 (PPCA)不仅关注数据的线性结构,还考虑了数据中的噪声 和缺失值 。概率主成分分析 (PPCA)的原理:其通过引入潜在变量来建模观测数据。其基本模型可以表示为:概率主成分分析 (PPCA)能够对每个数据点的分布进行建模:噪声 的方差。
概率主成分分析 (PPCA)的优势:
处理缺失值 :概率主成分分析 (PPCA)能够有效处理数据中的缺失值,而传统的主成分分析 (PCA)无法做到这一点。这使得概率主成分分析 (PPCA)在实际应用中更加灵活。概率模型 :由于概率主成分分析 (PPCA)基于概率模型 ,可以利用最大似然估计 (MLE)来估计参数,并进行统计推断。期望最大化算法 (EM算法):概率主成分分析 (PPCA)通常使用EM算法进行参数估计。EM算法通过迭代优化潜在变量和模型参数,提高了估计的准确性。
概率主成分分析 (PPCA)提供了一种更为灵活和强大的降维方法,通过引入概率模型 和潜在变量,它能够有效处理复杂的数据结构和缺失值问题。
独立成分分析(ICA) 独立成分分析 (ICA)是一种统计和计算技术,用于从多维信号中提取出相互独立的成分。独立成分分析 (ICA)特别适用于处理混合信号,尤其是在信号处理、图像处理和生物医学工程等领域。独立成分分析 (ICA)的原理是通过假设观测信号是若干个独立源信号的线性组合,来恢复这些源信号。其基本模型可以表示为:混合矩阵 ,表示源信号之间的线性组合。独立成分分析 (ICA)的目标是估计混合矩阵成分 尽可能独立。常用的独立成分分析 (ICA)算法包括:FastICA,一种迭代算法,通过最大化非高斯性 来估计独立成分,速度较快且易于实现;Infomax,基于信息论 的方法,通过最大化输出信息量来估计独立成分。独立成分分析 (ICA)是一种强大的工具,通过假设观测信号是多个独立源的线性组合,能够有效地从复杂数据中提取出有意义的信息。独立成分分析 (ICA)不是用于降低维度 的,而是用于分离叠加信号 。由于独立成分分析 (ICA)模型不包含噪声项,因此为了使模型正确,必须应用白化。
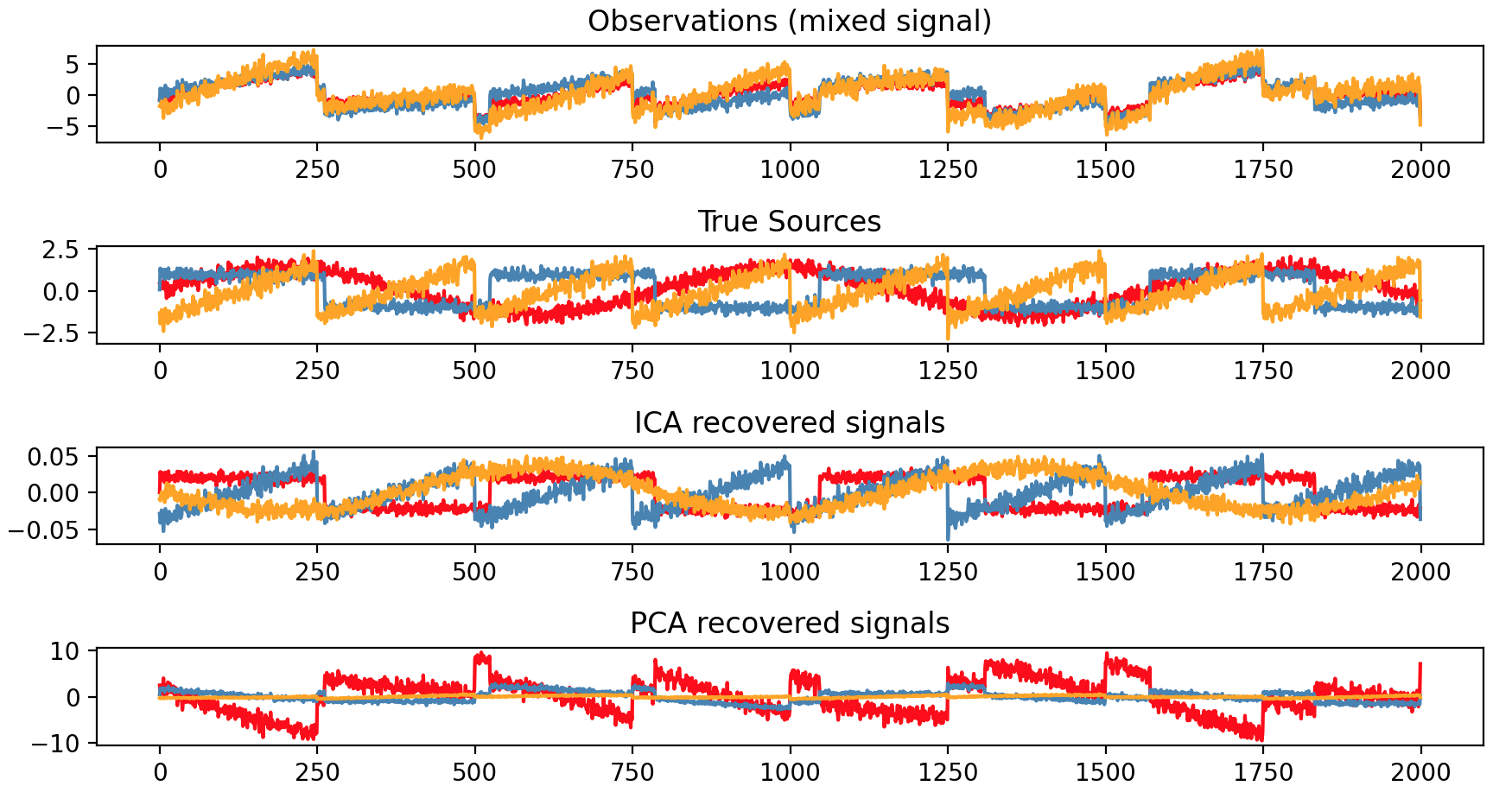
下面有一个例子是利用独立成分分析 (ICA)来估测噪声的来源。假设有3种乐器同时在演奏,3个麦克风录制混合信号。独立成分分析 (ICA)则用于恢复混合信号的来源,即每种乐器演奏的内容。因为相关信号(乐器)反映了非高斯过程,所以主成分分析 (PCA)无法恢复这些乐器信号。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 import numpy as npimport matplotlib.pyplot as pltfrom scipy import signalfrom sklearn.decomposition import PCA, FastICAnp.random.seed(0 ) n_samples = 2000 time = np.linspace(0 , 8 , n_samples) s1 = np.sin(2 * time) s2 = np.sign(np.sin(3 * time)) s3 = signal.sawtooth(2 * np.pi * time) S = np.c_[s1, s2, s3] S += 0.2 * np.random.normal(size=S.shape) S /= S.std(axis=0 ) A = np.array([[1 , 1 , 1 ], [0.5 , 2 , 1.0 ], [1.5 , 1.0 , 2.0 ]]) X = np.dot(S, A.T) ica = FastICA(n_components=3 , whiten="arbitrary-variance" ) S_ = ica.fit_transform(X) A_ = ica.mixing_ assert np.allclose(X, np.dot(S_, A_.T) + ica.mean_)pca = PCA(n_components=3 ) H = pca.fit_transform(X) plt.figure() models = [X, S, S_, H] names = ["Observations (mixed signal)" ,"True Sources" ,"ICA recovered signals" ,"PCA recovered signals" ,] colors = ["red" , "steelblue" , "orange" ] for ii, (model, name) in enumerate (zip (models, names), 1 ): plt.subplot(4 , 1 , ii) plt.title(name) for sig, color in zip (model.T, colors): plt.plot(sig, color=color) plt.tight_layout() plt.show()
非负矩阵分解(NMF) 非负矩阵分解 (NMF)是一种用于降维 和特征提取 的矩阵分解技术 ,广泛应用于信号处理、图像处理和文本挖掘等领域。非负矩阵分解 (NMF)的主要特点是要求矩阵的所有元素均为非负值,这使得其在许多实际应用中更具可解释性。非负矩阵分解 (NMF)的原理是将一个非负矩阵
在非负矩阵分解 (NMF)中,通常使用弗罗贝纽斯范数 (Frobenius norm)来衡量重构误差 。其目标函数可以表示为:弗罗贝纽斯范数 (Frobenius norm),定义为:非负矩阵分解 (NMF)的优化目标可以简化为:乘法更新法 ,通过迭代更新最小化重构误差 。更新规则如下:对于基矩阵0;交替最小二乘法 ,通过交替固定一个矩阵来优化另一个矩阵,以求解最优分解。使用弗罗贝纽斯范数 (Frobenius norm)的非负矩阵分解 (NMF)是一种有效的降维 和特征提取 方法,通过最小化重构误差 ,它能够提供高质量的结果。与PCA有所不同,向量的表示是通过叠加分量而不是减法方式获得的,而是以加法的方式获得。此类加法模型 对于表示图像和文本非常有效。
β散度 (Beta-Divergence)的非负矩阵分解 (NMF)是一种扩展的非负矩阵分解 (NMF)方法,旨在通过更灵活的损失函数 来捕捉数据的特性。β散度 提供了一种统一的框架,可以用于不同类型的数据分布,从而增强了非负矩阵分解 (NMF)在各种应用中的适用性。β散度 是一种衡量两个概率分布之间差异的度量,定义为:
当弗罗贝纽斯范数 (Frobenius norm),适用于高斯分布 。
当绝对误差 ,适用于拉普拉斯分布 。
当相对熵 (Kullback-Leibler散度),适用于泊松分布 。
在非负矩阵分解 (NMF)中,通过最小化
小批量非负矩阵分解 (Mini-batch NMF)是一种改进的非负矩阵分解 方法,旨在提高大规模数据集上非负矩阵分解 (NMF)的效率和可扩展性。通过使用小批量数据进行更新,这种方法能够显著减少计算时间和内存消耗,同时保持分解的质量。小批量非负矩阵分解 (Mini-batch NMF)的目标是将一个非负矩阵 非负矩阵 小批量非负矩阵分解 (Mini-batch NMF)中,整个数据集被划分为多个小批量,每个小批量包含一定数量的数据样本。每次迭代仅使用一个小批量来更新基矩阵 系数矩阵 小批量非负矩阵分解 (Mini-batch NMF)的执行步骤为:
数据分批 :将原始数据矩阵 初始化 :随机初始化基矩阵 系数矩阵 迭代更新 :对于每个小批量,进行以下步骤:1.计算当前小批量的重构误差 ;2.使用乘法更新法 或其他优化算法更新基矩阵 系数矩阵 迭代直到收敛 :重复上述步骤,直到达到预定的迭代次数或满足收敛条件。
小批量非负矩阵分解 (Mini-batch NMF)是一种高效且灵活的NMF方法,通过结合小批量处理技术,它不仅提高了训练速度,还增强了模型的可扩展性 和鲁棒性 。
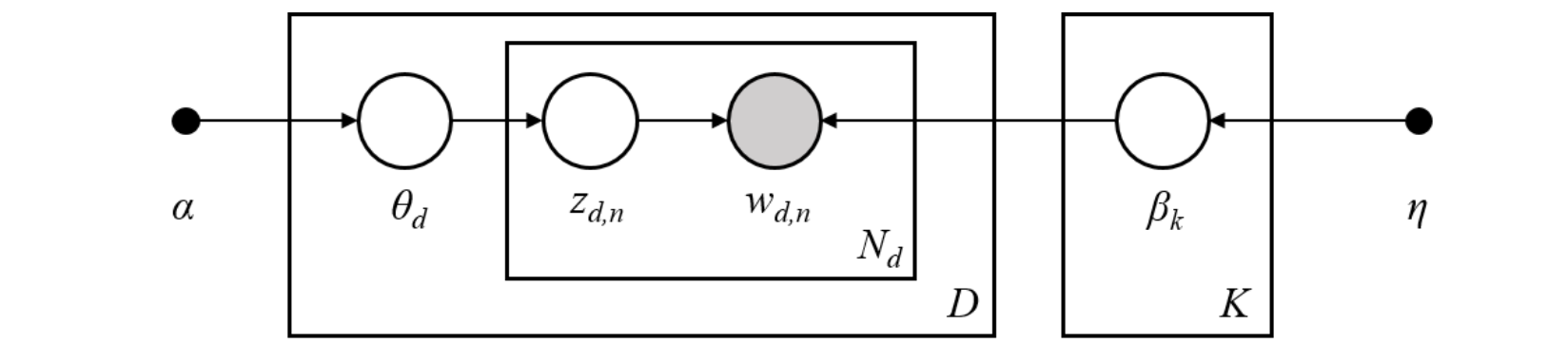
潜在狄利克雷分配(LDA) 潜在狄利克雷分配 (LDA)是一种广泛使用的主题模型,旨在从文档集合中发现潜在主题。潜在狄利克雷分配 (LDA)通过假设每个文档是由多个主题生成的,而每个主题又是由多个词构成的,从而实现对文本数据的建模和分析。潜在狄利克雷分配 (LDA)广泛应用于多个领域,包括文本挖掘、推荐系统、社交网络分析等领域。潜在狄利克雷分配 (LDA)的核心思想是将文档视为主题的混合,每个主题由一组词以不同的概率分布生成。其基本模型可以用以下步骤描述:主题分布 ,为每个文档生成一个主题分布,假设遵循狄利克雷分布 。具体来说,对于文档稀疏性 ;生成词 ,对于文档中的每个词:从主题分布狄利克雷分布 ;模型参数 ,潜在狄利克雷分配 (LDA)模型包括超参数 词分布参数
潜在狄利克雷分配 (LDA)的推断过程旨在估计给定文档集合下的隐含主题和相关参数。常用的方法包括:变分推断 (Variational Inference),通过优化变分下界来近似后验分布 ;吉布斯采样 (Gibbs Sampling),一种马尔可夫链蒙特卡罗 (MCMC)方法,通过迭代采样 来估计模型参数。潜在狄利克雷分配 (LDA)的优势包括:
可解释性 :潜在狄利克雷分配 (LDA)提供了清晰的主题表示,使得每个主题由一组相关词构成,便于理解和分析。灵活性 :能够处理大规模文本数据,并适用于多种文本挖掘任务,如文档聚类、推荐系统和信息检索。无监督学习 :潜在狄利克雷分配 (LDA)是一种无监督学习 方法,不需要预先标注的数据即可发现潜在结构。
潜在狄利克雷分配 (LDA)是一种强大的主题建模工具,通过假设文档由多个潜在主题生成,它能够有效地从大规模文本数据中提取有意义的信息。随着自然语言处理和机器学习的发展,潜在狄利克雷分配 (LDA)在许多实际应用中展现了其重要价值,尤其是在需要理解和分析文本内容时。
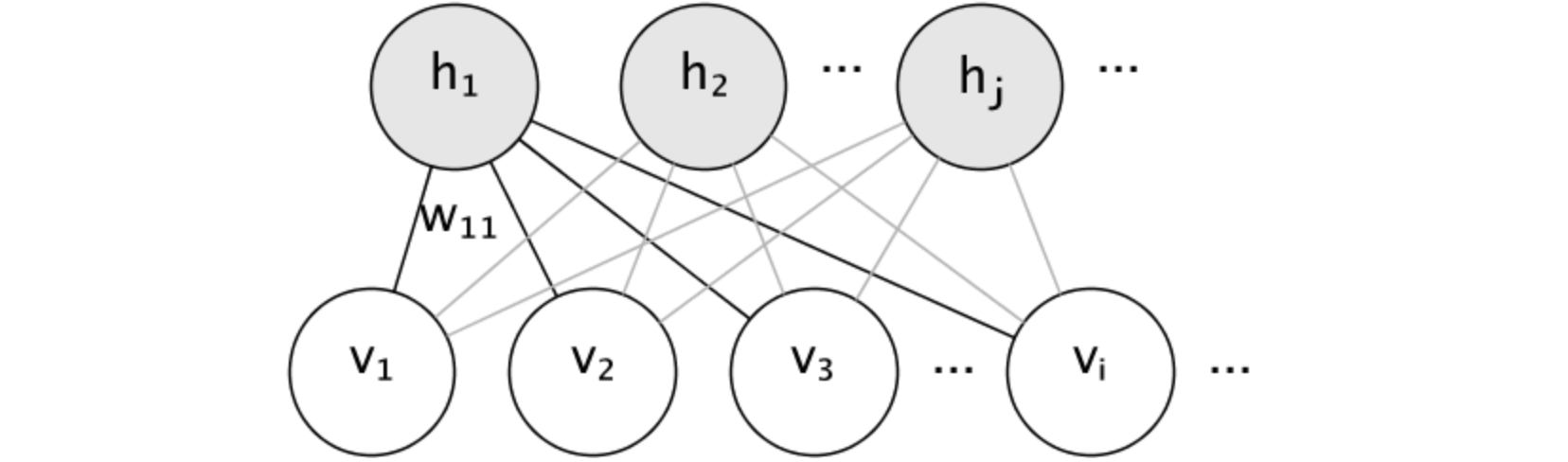
限制玻尔兹曼机(RBM) 限制玻尔兹曼机 (RBM)是一种无监督学习算法 ,属于深度学习中的生成模型。限制玻尔兹曼机 (RBM)由一组可见单元 (visible units)和一组隐藏单元 (hidden units)组成,二者之间通过权重连接,但同一层的单元之间没有连接。限制玻尔兹曼机 (RBM)广泛应用于多个领域,包括:推荐系统、图像处理、自然语言处理。限制玻尔兹曼机 (RBM)的结构可以用以下几个要素来描述:可见层 (Visible Layer):表示输入数据的特征,通常与观测数据直接对应。每个可见单元 表示一个特征或输入值。隐藏层 (Hidden Layer):用于捕捉输入数据中的潜在特征。隐藏单元 通过与可见单元 的连接来学习数据的隐含表示 。权重矩阵 (Weight Matrix):连接可见层 和隐藏层 的权重矩阵 可见单元 和隐藏单元 之间的关系。偏置项 (Bias Terms):每个可见单元 和隐藏单元 都有一个偏置项 ,用于调整激活值 。
限制玻尔兹曼机 (RBM)的工作原理是通过对输入数据进行概率建模来学习特征,其主要步骤如下:
前向传播 :给定可见单元的输入隐藏单元 的激活概率:激活函数 (sigmoid),隐藏单元 的偏置,可见单元 和隐藏单元 的权重。采样 :根据计算出的概率,从隐藏单元 中采样得到激活状态重构 :使用隐藏单元 的状态重构可见层:对比散度 (Contrastive Divergence):通过对比散度算法 更新权重 和偏置 ,以最小化输入数据和重构数据之间的差异。这一过程通常涉及多个迭代步骤。
限制玻尔兹曼机 (RBM)的优势:
无监督学习 :限制玻尔兹曼机 (RBM)能够在没有标签的数据上进行训练,适用于大规模未标注数据集。特征学习 :通过隐含层 捕捉数据中的潜在结构,使得限制玻尔兹曼机 (RBM)能够自动提取有用特征。生成能力 :限制玻尔兹曼机 (RBM)不仅可以用于特征提取,还可以生成新样本,具有良好的生成模型性能。
限制玻尔兹曼机 (RBM)是一种强大的无监督学习 工具,通过结合可见层 和隐藏层 ,它能够有效地捕捉数据中的潜在结构。
下面有一个示例,利用伯努利限制玻尔兹曼机模型 对灰度图像数据提取非线性特征,例如手写数字识别,为了从小数据集中学习到好的潜在表示,可以通过在每个方向上以1个像素的线性移位扰动训练数据来生成更多标记数据:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 import numpy as npimport matplotlib.pyplot as pltfrom sklearn import metricsfrom sklearn import datasetsfrom sklearn.base import clonefrom scipy.ndimage import convolvefrom sklearn.model_selection import train_test_splitfrom sklearn.preprocessing import minmax_scalefrom sklearn import linear_modelfrom sklearn.neural_network import BernoulliRBMfrom sklearn.pipeline import Pipelinedef nudge_dataset (X, Y ): """ This produces a dataset 5 times bigger than the original one, by moving the 8x8 images in X around by 1px to left, right, down, up """ direction_vectors = [ [[0 , 1 , 0 ], [0 , 0 , 0 ], [0 , 0 , 0 ]], [[0 , 0 , 0 ], [1 , 0 , 0 ], [0 , 0 , 0 ]], [[0 , 0 , 0 ], [0 , 0 , 1 ], [0 , 0 , 0 ]], [[0 , 0 , 0 ], [0 , 0 , 0 ], [0 , 1 , 0 ]], ] def shift (x, w ): return convolve(x.reshape((8 , 8 )), mode="constant" , weights=w).ravel() X = np.concatenate([X] + [np.apply_along_axis(shift, 1 , X, vector) for vector in direction_vectors]) Y = np.concatenate([Y for _ in range (5 )], axis=0 ) return X, Y X, y = datasets.load_digits(return_X_y=True ) X = np.asarray(X, "float32" ) X, Y = nudge_dataset(X, y) X = minmax_scale(X, feature_range=(0 , 1 )) X_train, X_test, Y_train, Y_test = train_test_split(X, Y, test_size=0.2 , random_state=0 ) logistic = linear_model.LogisticRegression(solver="newton-cg" , tol=1 ) rbm = BernoulliRBM(random_state=0 , verbose=True ) rbm_features_classifier = Pipeline(steps=[("rbm" , rbm), ("logistic" , logistic)]) rbm.learning_rate = 0.06 rbm.n_iter = 10 rbm.n_components = 100 logistic.C = 6000 rbm_features_classifier.fit(X_train, Y_train) raw_pixel_classifier = clone(logistic) raw_pixel_classifier.C = 100.0 raw_pixel_classifier.fit(X_train, Y_train) Y_pred = rbm_features_classifier.predict(X_test) print ("Logistic regression using RBM features:\n%s\n" % (metrics.classification_report(Y_test, Y_pred)))plt.figure(figsize=(4.2 , 4 )) for i, comp in enumerate (rbm.components_): plt.subplot(10 , 10 , i + 1 ) plt.imshow(comp.reshape((8 , 8 )), cmap=plt.cm.gray_r, interpolation="nearest" ) plt.xticks(()) plt.yticks(()) plt.suptitle("100 components extracted by RBM" , fontsize=16 ) plt.subplots_adjust(0.08 , 0.02 , 0.92 , 0.85 , 0.08 , 0.23 ) plt.show()
输出评估结果为:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 Logistic regression using RBM features: precision recall f1-score support 0 0.10 1.00 0.18 174 1 0.00 0.00 0.00 184 2 0.00 0.00 0.00 166 3 0.00 0.00 0.00 194 4 0.00 0.00 0.00 186 5 0.00 0.00 0.00 181 6 0.00 0.00 0.00 207 7 0.00 0.00 0.00 154 8 0.00 0.00 0.00 182 9 0.00 0.00 0.00 169 accuracy 0.10 1797 macro avg 0.01 0.10 0.02 1797 weighted avg 0.01 0.10 0.02 1797
图模型与参数化 限制玻尔兹曼机 (RBM)的图模型是全连通的二分图。
节点是随机变量,其状态取决于它们所连接的其他节点的状态。因此,该模型由连接的权重以及每个可见 和隐藏单元 的一个截距 (偏差)项参数化,为简单起见,图像中省略了该项。能量函数衡量联合分配的质量:可见层 和隐藏层 的截距向量 。模型的联合概率 根据能量定义:二分结构 ,它禁止隐藏单元 之间或可见单元 之间的直接交互。意味着:块吉布斯采样 (Block Gibbs Sampling)进行推理。
能量函数 (Energy Function)是一个数学表示,用于量化 系统某一状态所具有的能量。它在多个领域中具有重要应用,包括物理学、工程学和优化算法。能量函数 (Energy Function)用于描述系统的状态,并评估该状态的可行性或优越性。在优化问题中,能量函数 (Energy Function)通常用于指导搜索算法,如模拟退火 和禁忌搜索 ,通过提供一个度量来判断解的接近程度,从而影响接受或拒绝新候选解的决策。在优化问题中,能量函数 (Energy Function)用于评估不同解的优劣。例如,在机器学习和计算机视觉中,能量函数 (Energy Function)可以帮助模型找到最优参数或配置。通过最小化能量函数,可以找到最优解或近似最优解。
块吉布斯采样 (Block Gibbs Sampling)是一种马尔可夫链蒙特卡罗 (MCMC)方法,用于从复杂的多维概率分布中抽样。与传统的吉布斯采样 不同,块吉布斯采样 (Block Gibbs Sampling)在每次迭代中同时更新多个变量,而不是逐个更新。这种方法在处理高维数据和具有复杂依赖关系的模型时特别有效。
伯努利限制玻尔兹曼机 伯努利限制玻尔兹曼机 (BRBM)是一种特殊类型的限制玻尔兹曼机 (RBM),其中可见层 的单元使用伯努利分布 来建模。这使得伯努利限制玻尔兹曼机 (BRBM)特别适用于处理二元(0-1)数据,如图像的黑白像素或用户偏好(喜欢/不喜欢)等。其模型结构与工作原理都与限制玻尔兹曼机 (RBM)相同。伯努利限制玻尔兹曼机 (BRBM)优势:
适用于二元数据 :伯努利限制玻尔兹曼机 (BRBM)特别适合处理二进制或稀疏数据,使其在许多实际应用中表现良好。特征学习 :通过隐含层捕捉数据中的潜在结构,使得伯努利限制玻尔兹曼机 (BRBM)能够自动提取有用特征。生成能力 :伯努利限制玻尔兹曼机 (BRBM)不仅可以用于特征提取,还可以生成新样本,具有良好的生成模型性能。
计算隐藏单元 的激活概率:激活函数 (sigmoid),隐藏单元 的偏置,可见单元 和隐藏单元 的权重。使用隐藏单元 的状态重构可见层:
随机最大似然学习 随机最大似然学习 (Stochastic Maximum Likelihood Learning)是一种用于参数估计的优化方法,特别适用于处理大规模数据集和复杂模型。它结合了最大似然估计 (MLE)和随机梯度下降 (Stochastic Gradient Descent, SGD)等技术,以提高学习效率和收敛速度。随机最大似然学习 的应用领域包括:机器学习、信号处理、生物信息学等。
随机最大似然学习 (Stochastic Maximum Likelihood Learning)是一种统计方法,用于估计模型参数,使得在给定数据的情况下,观察到的数据具有最高的概率。对于给定的参数最大似然估计 的目标是最大化似然函数 :
随机梯度下降 是一种优化算法,通过使用小批量(mini-batch)或单个样本来更新模型参数,从而减少计算成本并加速收敛。其基本步骤如下:1.从数据集中随机选择一个样本或小批量;2.计算损失函数 的梯度 ;3.更新参数:随机最大似然学习 的步骤:
初始化参数 :随机初始化模型参数。选择小批量 :从训练数据中随机选择一个小批量样本。计算梯度 :根据当前参数计算小批量样本的似然函数的梯度。更新参数 :使用计算得到的梯度更新模型参数。迭代 :重复上述步骤,直到达到预定的迭代次数或满足收敛条件。
随机最大似然学习 的优势:高效性 ,通过使用小批量样本进行更新,随机最大似然学习 能够显著减少每次迭代所需的计算时间,适合大规模数据集;收敛速度快 ,由于引入了随机性,能够避免陷入局部最优解,从而提高收敛速度;适应性强 ,可以灵活应用于各种模型,包括线性回归 、神经网络 和隐马尔可夫模型 等。随机最大似然学习 是一种高效的参数估计方法,通过结合最大似然估计 和随机梯度下降 ,它能够在大规模数据集上实现快速收敛。
协方差估计(Covariance Estimation) 协方差估计 (Covariance Estimation)是统计学中用于估计随机变量之间的协方差 的过程。协方差 是衡量两个随机变量之间关系的一个重要指标,反映了它们如何共同变化。准确估计协方差 对于许多应用至关重要,包括金融建模、机器学习和信号处理等。许多统计问题都需要估计总体的协方差矩阵 ,这可以看作是对数据集散点图形状的估计。大多数情况下,这种估计必须对样本进行,样本的属性(大小、结构、同质性)对估计的质量有很大影响。协方差 定义:给定两个随机变量期望值 。协方差矩阵 定义:对于多个随机变量,可以构建一个协方差矩阵
协方差估计 (Covariance Estimation)方法有:
样本协方差矩阵 (Sample Covariance Matrix):样本协方差矩阵 是最常用的估计方法,适用于完整数据集。其计算公式为:正则化和收缩方法 (Shrinkage Methods):在高维数据中,样本协方差矩阵 可能会不稳定或非正定。收缩方法通过将样本协方差矩阵 向某个目标矩阵(如单位矩阵)收缩,从而提高估计的稳定性和准确性。图模型方法 (Graphical Models):图模型 (如高斯图模型 )直接估计精度矩阵 (协方差矩阵 的逆),在某些情况下比直接估计协方差矩阵 更有效。自适应方法 (Adaptive Methods):这些方法根据数据特性自适应地调整估计过程,以提高准确性。例如,使用加权平均 或局部回归技术 来估计协方差 。
协方差估计 (Covariance Estimation)是统计分析中的关键步骤,涉及多种方法和技术。选择合适的估计方法取决于数据的特性、维度以及应用场景。通过精确的协方差估计 ,可以更好地理解变量之间的关系,并做出更有效的决策。
经验协方差 经验协方差 (Empirical Covariance)是通过样本数据计算出的协方差 ,用于估计总体协方差 。它是描述两个随机变量之间关系的重要统计量,能够反映这两个变量在样本中的共同变化程度。经验协方差 (Empirical Covariance)定义:经验协方差 用于衡量两个随机变量计算样本均值 ,首先计算每个变量的样本均值;计算偏差 ,对于每一对观测值,计算它们与各自均值的偏差;2.求积并求和 ,将偏差相乘并累加;3.归一化 ,将总和除以经验协方差 。经验方差 的特点是:
正负符号 :正协方差 ,表示两个变量同向变化,即一个变量增加时另一个变量也增加;负协方差 ,表示两个变量反向变化,即一个变量增加时另一个变量减少。数值范围 :经验协方差 的数值没有上限或下限,可以是正数、负数或零。零协方差 表示两个变量之间没有线性关系。
经验协方差 是通过样本数据计算出的重要统计量,能够有效地描述和量化 随机变量之间的关系。
收缩协方差(Shrunk Covariance) 收缩协方差 (Shrunk Covariance)是一种用于提高协方差矩阵估计 稳定性和准确性的技术,特别是在高维数据分析中。传统的样本协方差矩阵 在样本量相对较小或变量数量较多时,可能会变得不稳定或非正定(即其特征值中存在负值),这会影响后续的统计分析和模型构建。收缩协方差 (Shrunk Covariance)的工作原理是:收缩协方差 通过将样本协方差矩阵 向某个目标矩阵 (通常是单位矩阵 或对角矩阵 )收缩,从而减少估计的方差,提高估计的可靠性。其基本思想是结合样本协方差 和目标矩阵 ,形成一个新的协方差估计 。它的数学表示:设样本协方差矩阵 ,目标协方差矩阵 (如单位矩阵)收缩后的协方差矩阵[0,1]之间,通过调整样本协方差 和目标协方差 之间的权重。
收缩协方差 (Shrunk Covariance)的优势:
提高稳定性 :通过引入目标矩阵 ,收缩协方差 能够减少样本协方差 的波动性,使得估计更加稳定。防止非正定 :在高维情况下,样本协方差 可能会变得非正定,而收缩方法可以确保得到一个正定矩阵,从而适用于后续的分析。适应性强 :根据数据特性自适应地调整收缩程度,可以有效提高模型性能。
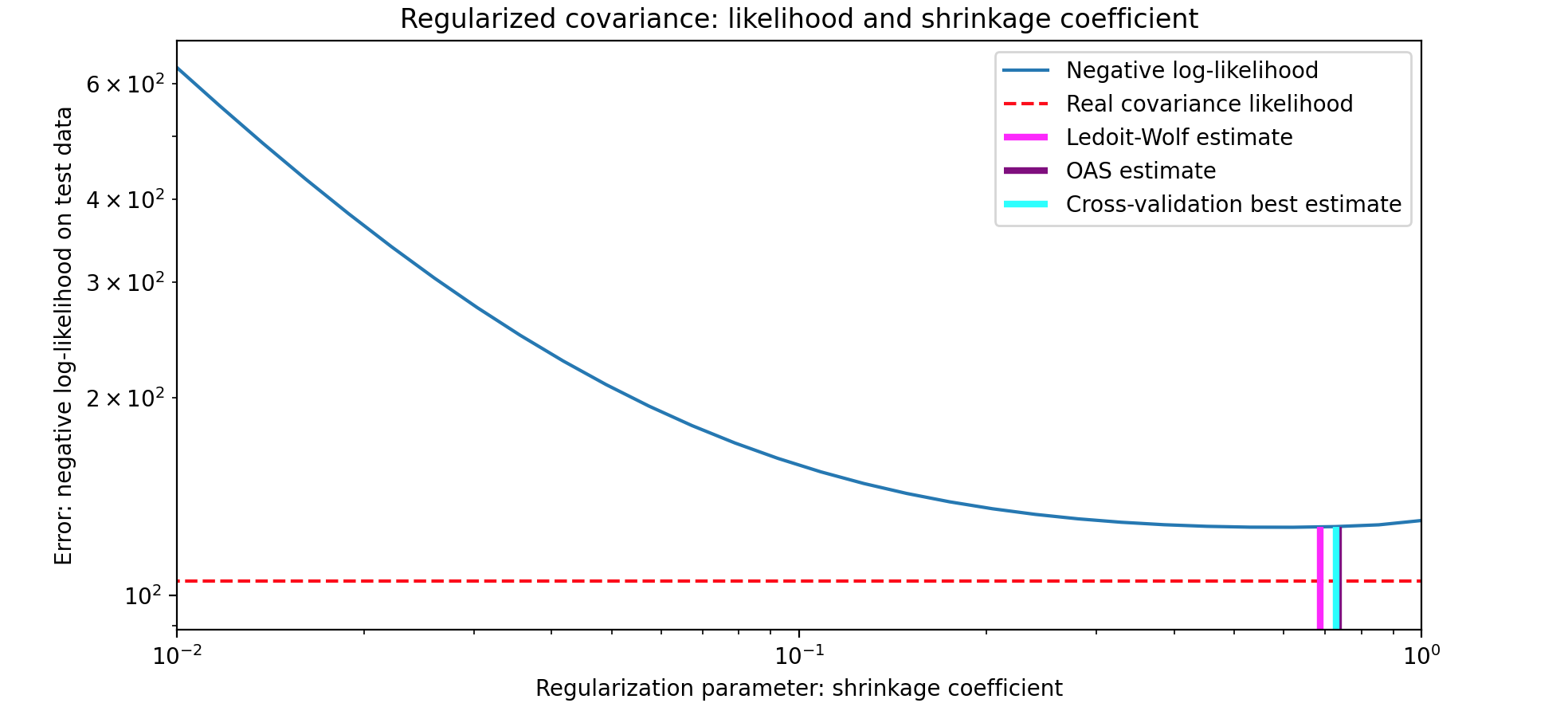
收缩协方差 (Shrunk Covariance)方法示例有:Ledoit-Wolf收缩,一种常用的收缩方法,通过最小化均方误差 来选择最优的收缩参数对角收缩 :将样本协方差收缩到对角矩阵 ,以简化模型并减少复杂性。下面有一个示例,利用收缩协方差 估计量中使用的正则化来提高协方差 的准确性。这里比较了三种方法:Ledoit-Wolf(通过最小化均方误差 来选择最优的收缩参数OAS(假设数据呈现高斯分布,其收敛性明显更好,尤其是对于小样本)和最大似然估计。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 import numpy as npimport matplotlib.pyplot as pltfrom scipy import linalgfrom sklearn.covariance import OAS, LedoitWolffrom sklearn.model_selection import GridSearchCVfrom sklearn.covariance import ShrunkCovariance, empirical_covariance, log_likelihoodn_features, n_samples = 40 , 20 np.random.seed(42 ) base_X_train = np.random.normal(size=(n_samples, n_features)) base_X_test = np.random.normal(size=(n_samples, n_features)) coloring_matrix = np.random.normal(size=(n_features, n_features)) X_train = np.dot(base_X_train, coloring_matrix) X_test = np.dot(base_X_test, coloring_matrix) shrinkages = np.logspace(-2 , 0 , 30 ) negative_logliks = [-ShrunkCovariance(shrinkage=s).fit(X_train).score(X_test) for s in shrinkages] real_cov = np.dot(coloring_matrix.T, coloring_matrix) emp_cov = empirical_covariance(X_train) loglik_real = -log_likelihood(emp_cov, linalg.inv(real_cov)) tuned_parameters = [{"shrinkage" : shrinkages}] cv = GridSearchCV(ShrunkCovariance(), tuned_parameters) cv.fit(X_train) lw = LedoitWolf() loglik_lw = lw.fit(X_train).score(X_test) oa = OAS() loglik_oa = oa.fit(X_train).score(X_test) fig = plt.figure() plt.title("Regularized covariance: likelihood and shrinkage coefficient" ) plt.xlabel("Regularization parameter: shrinkage coefficient" ) plt.ylabel("Error: negative log-likelihood on test data" ) plt.loglog(shrinkages, negative_logliks, label="Negative log-likelihood" ) plt.plot(plt.xlim(), 2 * [loglik_real], "--r" , label="Real covariance likelihood" ) lik_max = np.amax(negative_logliks) lik_min = np.amin(negative_logliks) ymin = lik_min - 6.0 * np.log((plt.ylim()[1 ] - plt.ylim()[0 ])) ymax = lik_max + 10.0 * np.log(lik_max - lik_min) xmin = shrinkages[0 ] xmax = shrinkages[-1 ] plt.vlines(lw.shrinkage_, ymin, -loglik_lw, color="magenta" , linewidth=3 , label="Ledoit-Wolf estimate" ,) plt.vlines(oa.shrinkage_, ymin, -loglik_oa, color="purple" , linewidth=3 , label="OAS estimate" ) plt.vlines(cv.best_estimator_.shrinkage, ymin, -cv.best_estimator_.score(X_test), color="cyan" , linewidth=3 , label="Cross-validation best estimate" ,) plt.ylim(ymin, ymax) plt.xlim(xmin, xmax) plt.legend() plt.show()
稀疏逆协方差(Sparse Inverse Covariance) 稀疏逆协方差 (Sparse Inverse Covariance)是一种用于估计高维数据中的协方差矩阵逆 (精度矩阵 )的方法,尤其适用于变量数量远大于样本数量的情况。通过引入稀疏性约束 ,这种方法能够有效地识别变量之间的条件独立性,从而提供更具可解释性的模型。协方差矩阵 :描述多个随机变量之间的线性关系。逆协方差矩阵 :是协方差矩阵 的逆,反映了变量之间的条件独立性。若协方差矩阵 ,则其逆为稀疏性 :在高维数据中,许多变量可能是条件独立的。稀疏逆协方差估计 通过强制大多数元素为0,来简化逆协方差矩阵 ,从而使得模型更易于解释和分析。
稀疏逆协方差 (Sparse Inverse Covariance)估计方法:
Lasso方法:稀疏逆协方差估计 通常使用Lasso回归(L1正则化)来实现。目标是最小化以下损失函数 :样本协方差矩阵 ,逆协方差矩阵 ,稀疏性 。Graphical Lasso方法:Graphical Lasso是一种具体实现,它利用优化算法(如坐标下降法)来求解上述优化问题。通过迭代更新,可以有效地获得稀疏逆协方差估计 。
稀疏逆协方差 (Sparse Inverse Covariance)优势:
可解释性 :通过引入稀疏性 ,能够明确识别变量之间的关系,提供更清晰的模型解释。处理高维数据 :在变量数量远大于样本数量的情况下,稀疏逆协方差估计能够有效避免传统方法的不稳定性。条件独立性推断 :能够揭示变量之间的条件独立结构,为构建图形模型(如贝叶斯网络 )提供支持。
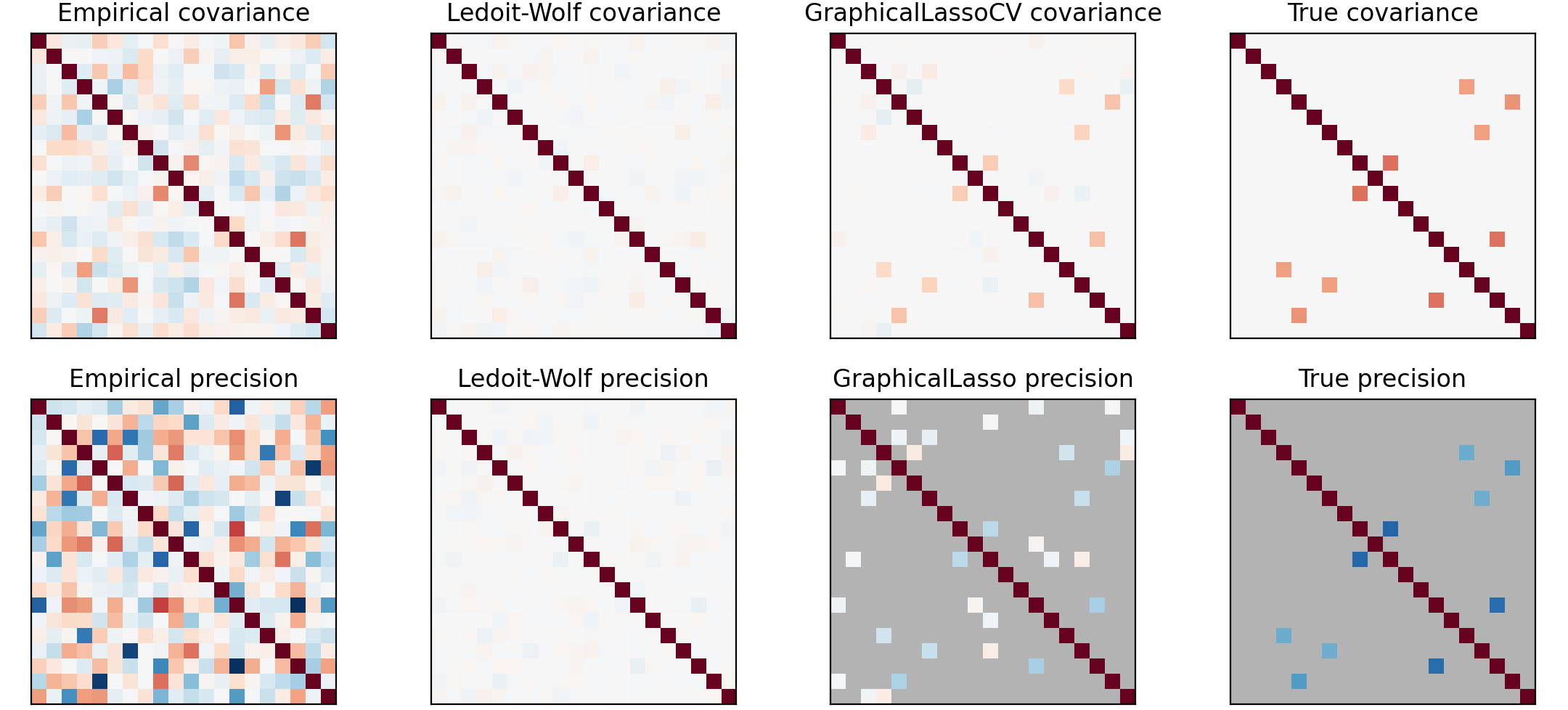
这里有一个协方差估计和稀疏精度的实例,高斯模型 是由协方差矩阵逆 (精度矩阵 )参数化的。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 import numpy as npimport matplotlib.pyplot as pltfrom scipy import linalgfrom sklearn.datasets import make_sparse_spd_matrixfrom sklearn.covariance import GraphicalLassoCV, ledoit_wolfn_samples = 60 n_features = 20 prng = np.random.RandomState(1 ) prec = make_sparse_spd_matrix(n_features, alpha=0.98 , smallest_coef=0.4 , largest_coef=0.7 , random_state=prng) cov = linalg.inv(prec) d = np.sqrt(np.diag(cov)) cov /= d cov /= d[:, np.newaxis] prec *= d prec *= d[:, np.newaxis] X = prng.multivariate_normal(np.zeros(n_features), cov, size=n_samples) X -= X.mean(axis=0 ) X /= X.std(axis=0 ) emp_cov = np.dot(X.T, X) / n_samples model = GraphicalLassoCV() model.fit(X) cov_ = model.covariance_ prec_ = model.precision_ lw_cov_, _ = ledoit_wolf(X) lw_prec_ = linalg.inv(lw_cov_) plt.figure(figsize=(10 , 6 )) plt.subplots_adjust(left=0.02 , right=0.98 ) covs = [("Empirical" , emp_cov),("Ledoit-Wolf" , lw_cov_),("GraphicalLassoCV" , cov_),("True" , cov),] vmax = cov_.max () for i, (name, this_cov) in enumerate (covs): plt.subplot(2 , 4 , i + 1 ) plt.xticks(()) plt.yticks(()) plt.title("%s covariance" % name) plt.imshow(this_cov, interpolation="nearest" , vmin=-vmax, vmax=vmax, cmap=plt.cm.RdBu_r) precs = [("Empirical" , linalg.inv(emp_cov)),("Ledoit-Wolf" , lw_prec_),("GraphicalLasso" , prec_),("True" , prec),] vmax = 0.9 * prec_.max () for i, (name, this_prec) in enumerate (precs): ax = plt.subplot(2 , 4 , i + 5 ) plt.xticks(()) plt.yticks(()) plt.imshow(np.ma.masked_equal(this_prec, 0 ),interpolation="nearest" ,vmin=-vmax,vmax=vmax,cmap=plt.cm.RdBu_r,) plt.title("%s precision" % name) if hasattr (ax, "set_facecolor" ): ax.set_facecolor(".7" ) else : ax.set_axis_bgcolor(".7" ) plt.show()
鲁棒协方差估计 (Robust Covariance Estimation)鲁棒协方差估计 (Robust Covariance Estimation)是一种用于估计协方差矩阵 的方法,旨在提高对异常值和噪声的抵抗能力。传统的协方差估计 方法(如样本协方差 )在数据中存在异常值时可能会产生偏差,导致不准确的结果。鲁棒协方差估计 通过引入特定的技术和算法,能够更好地处理这些问题。异常值 :异常值 是指与其他数据点显著不同的观测值,它们可能由于测量错误、数据输入错误或自然变异而出现。在统计分析中,异常值 会对均值 和协方差 等统计量产生重大影响。鲁棒协方差估计 应用领域包括:金融分析、生物统计学、机器学习等。
鲁棒协方差估计 (Robust Covariance Estimation)的方法:
M估计 (M-Estimators):M估计是一种广泛使用的方法,通过最小化某个损失函数 来获得参数估计。对于鲁棒协方差估计 ,可以使用加权最小二乘法 来降低异常值对结果的影响。最小绝对偏差 (MAD):使用绝对偏差 而不是平方偏差 来计算中心位置和散布程度 ,从而减少异常值的影响。Huber损失函数 :Huber损失函数结合了平方损失 和绝对损失 ,能够在异常值存在时提供更鲁棒的估计。它在一定范围内使用平方损失,而在超出范围时使用绝对损失。最小化分位数 (Quantile Minimization):通过最小化数据分位数 来获得鲁棒的参数估计,从而减少异常值的影响。S-estimator和T-estimator:S-estimator基于样本散布进行鲁棒估计 ,而T-estimator则基于样本均值和样本散布进行鲁棒估计 。
鲁棒协方差估计 (Robust Covariance Estimation)的优势:
抵御异常值 :鲁棒协方差估计 能够有效地减少异常值对结果的影响,从而提供更可靠的参数估计。适用性强 :适用于各种类型的数据,包括高维数据和有噪声的数据集。提高模型性能 :通过提供更准确的协方差矩阵 ,鲁棒协方差估计 能够改善后续分析和建模的结果。
鲁棒协方差估计 (Robust Covariance Estimation)是一种重要的统计工具,通过引入特定的方法和技术,有效地处理数据中的异常值和噪声。它在多个领域中展现了其重要价值,为理解复杂系统中的变量关系提供了可靠的方法。最小协方差行列式 (MCD)是一种鲁棒协方差估计 的方法,旨在从数据集中识别和排除异常值,从而提供更可靠的协方差矩阵估计 。MCD方法特别适用于高维数据分析,能够有效地处理存在异常值的情况。最小协方差行列式 (MCD)方法通过寻找一个子集,使得该子集的协方差行列式 最小化,从而获得鲁棒协方差估计 (Robust Covariance Estimation)。具体步骤如下:
选择子集 :从数据集中选择计算协方差矩阵 :计算该子集的样本协方差矩阵 。最小化行列式 :最小化所选子集的协方差矩阵 的行列式,即:协方差矩阵 。重复迭代 :通过不同的组合不断重复上述步骤,直到找到使得行列式最小的最佳子集。
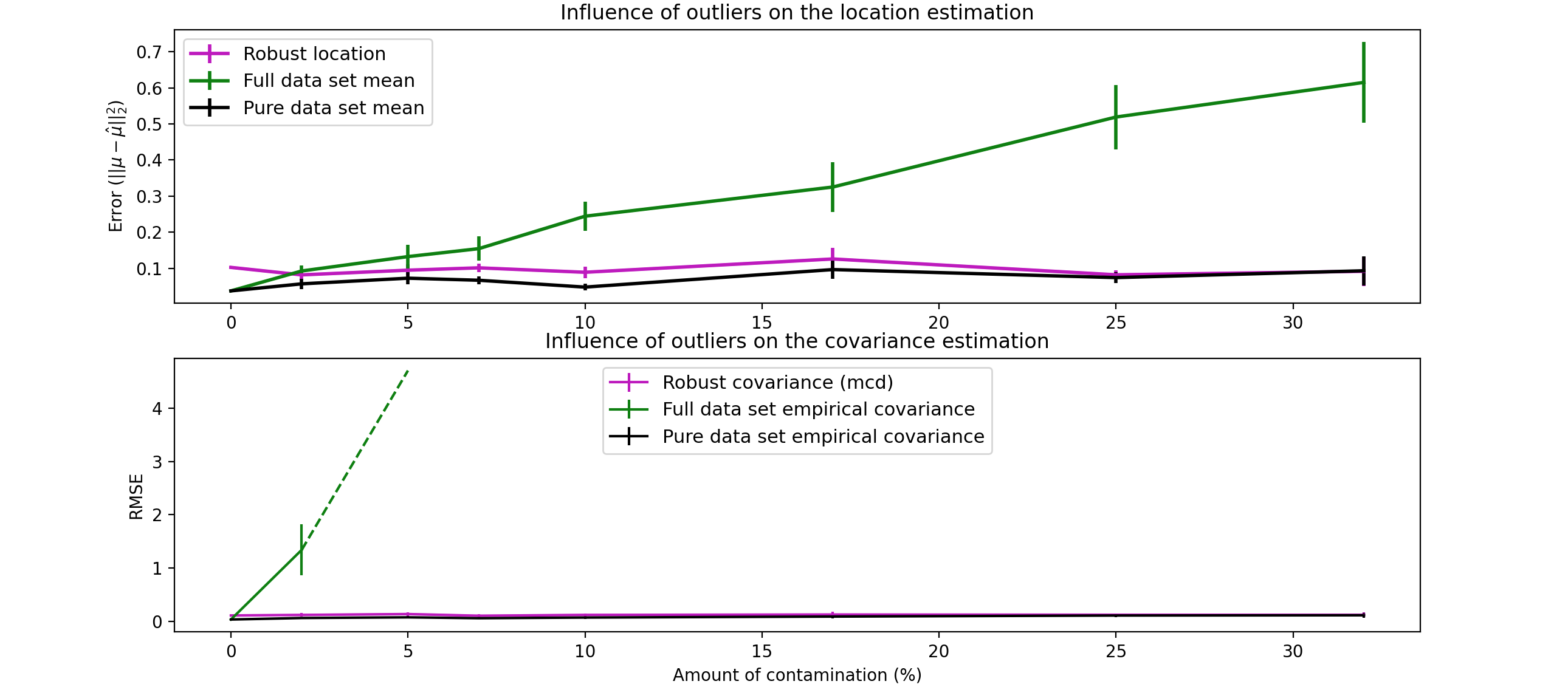
下面有一个示例,比较了在受污染的高斯分布数据集上使用各种类型(数据集平均值 和协方差 )的协方差估计 (鲁棒协方差 (MCD)和经验协方差 )时产生的估计误差 :
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 import matplotlib.font_managerimport matplotlib.pyplot as pltimport numpy as npfrom sklearn.covariance import EmpiricalCovariance, MinCovDetn_samples = 80 n_features = 5 repeat = 10 range_n_outliers = np.concatenate((np.linspace(0 , n_samples / 8 , 5 ),np.linspace(n_samples / 8 , n_samples / 2 , 5 )[1 :-1 ],)).astype(int ) err_loc_mcd = np.zeros((range_n_outliers.size, repeat)) err_cov_mcd = np.zeros((range_n_outliers.size, repeat)) err_loc_emp_full = np.zeros((range_n_outliers.size, repeat)) err_cov_emp_full = np.zeros((range_n_outliers.size, repeat)) err_loc_emp_pure = np.zeros((range_n_outliers.size, repeat)) err_cov_emp_pure = np.zeros((range_n_outliers.size, repeat)) for i, n_outliers in enumerate (range_n_outliers): for j in range (repeat): rng = np.random.RandomState(i * j) X = rng.randn(n_samples, n_features) outliers_index = rng.permutation(n_samples)[:n_outliers] outliers_offset = 10.0 * (np.random.randint(2 , size=(n_outliers, n_features)) - 0.5 ) X[outliers_index] += outliers_offset inliers_mask = np.ones(n_samples).astype(bool ) inliers_mask[outliers_index] = False mcd = MinCovDet().fit(X) err_loc_mcd[i, j] = np.sum (mcd.location_**2 ) err_cov_mcd[i, j] = mcd.error_norm(np.eye(n_features)) err_loc_emp_full[i, j] = np.sum (X.mean(0 ) ** 2 ) err_cov_emp_full[i, j] = (EmpiricalCovariance().fit(X).error_norm(np.eye(n_features))) pure_X = X[inliers_mask] pure_location = pure_X.mean(0 ) pure_emp_cov = EmpiricalCovariance().fit(pure_X) err_loc_emp_pure[i, j] = np.sum (pure_location**2 ) err_cov_emp_pure[i, j] = pure_emp_cov.error_norm(np.eye(n_features)) lw = 2 plt.subplot(2 , 1 , 1 ) font_prop = matplotlib.font_manager.FontProperties(size=11 ) plt.errorbar(range_n_outliers,err_loc_mcd.mean(1 ),yerr=err_loc_mcd.std(1 ) / np.sqrt(repeat),label="Robust location" ,lw=lw,color="m" ,) plt.errorbar(range_n_outliers,err_loc_emp_full.mean(1 ),yerr=err_loc_emp_full.std(1 ) / np.sqrt(repeat),label="Full data set mean" ,lw=lw,color="green" ,) plt.errorbar(range_n_outliers,err_loc_emp_pure.mean(1 ),yerr=err_loc_emp_pure.std(1 ) / np.sqrt(repeat),label="Pure data set mean" ,lw=lw,color="black" ,) plt.title("Influence of outliers on the location estimation" ) plt.ylabel(r"Error ($||\mu - \hat{\mu}||_2^2$)" ) plt.legend(loc="upper left" , prop=font_prop) plt.subplot(2 , 1 , 2 ) x_size = range_n_outliers.size plt.errorbar(range_n_outliers,err_cov_mcd.mean(1 ),yerr=err_cov_mcd.std(1 ),label="Robust covariance (mcd)" ,color="m" ,) plt.errorbar(range_n_outliers[: (x_size // 5 + 1 )],err_cov_emp_full.mean(1 )[: (x_size // 5 + 1 )],yerr=err_cov_emp_full.std(1 )[: (x_size // 5 + 1 )], label="Full data set empirical covariance" ,color="green" ) plt.plot(range_n_outliers[(x_size // 5 ) : (x_size // 2 - 1 )],err_cov_emp_full.mean(1 )[(x_size // 5 ) : (x_size // 2 - 1 )],color="green" ,ls="--" ,) plt.errorbar(range_n_outliers,err_cov_emp_pure.mean(1 ),yerr=err_cov_emp_pure.std(1 ),label="Pure data set empirical covariance" ,color="black" ,) plt.title("Influence of outliers on the covariance estimation" ) plt.xlabel("Amount of contamination (%)" ) plt.ylabel("RMSE" ) plt.legend(loc="upper center" , prop=font_prop) plt.show()